近日,脑科学与康复医学研究院梁九兴博士与中山大学附属第一医院胡争教授课题组合作,在数学与计算生物学领域国际知名期刊Briefings in Bioinformatics(2019年影响因子:8.99)在线发表了题为 “ DeepHPV: a deep learning model to predict human papillomavirus integration sites ”的研究论文(doi: 10.1093/bib/bbaa242)。
人乳头瘤病毒(HPV)是一种双链DNA病毒,在全球引起约4.5%的癌症,具体包括宫颈癌、肛门癌、阴道癌、阴茎癌、头颈癌等。病毒整合到人类宿主基因是癌症进展的重要演进步骤,进而导致对宿主细胞的损伤甚至癌变。关于人类基因组中的插入诱变是完全随机还是非随机的争论持续了数十年。近年来,越来越多的证据表明,HPV倾向于整合到宿主基因的特定区域,并为其生存选择发挥了生长优势。然而,HPV的整合并不是本身自然的生命延续的过程,这一过程比逆转录病毒的整合更加复杂,后者仅需处理特定的整合酶便可促进这一过程。因此,与逆转录病毒(如HIV)相比,对HPV整合模式的预测更加困难。
该研究为了能够准确地预测HPV在人类宿主DNA整合位点。本研究提出并开发了一种基于“注意力”机制的深度学习模型DeepHPV,通过自动提取特征并预测HPV整合位点。具体采用3608个已打标的HPV整合位点的DNA序列样本训练模型,并使用584个已打标的HPV整合位点的DNA序列样本作为测试数据集。DeepHPV的接收者操作特征曲线下面积(AUROC)为0.6336,精确率-召回率曲线下面积(AUPR)为0.5670。添加RepeatMasker和TCGA泛癌基因组学特征后模型的AUROC性能分别提高至0.8464和0.8501,AUPR性能分别提高到0.7985和0.8106。随后,我们在独立的数据库VISDB上测试了这些训练模型,发现添加TCGA泛癌特征的模型比添加RepeatMasker特征的模型(AUROC:0.6102,AUPR:0.5577)性能表现更佳(AUROC:0.7175,AUPR:0.6284)。
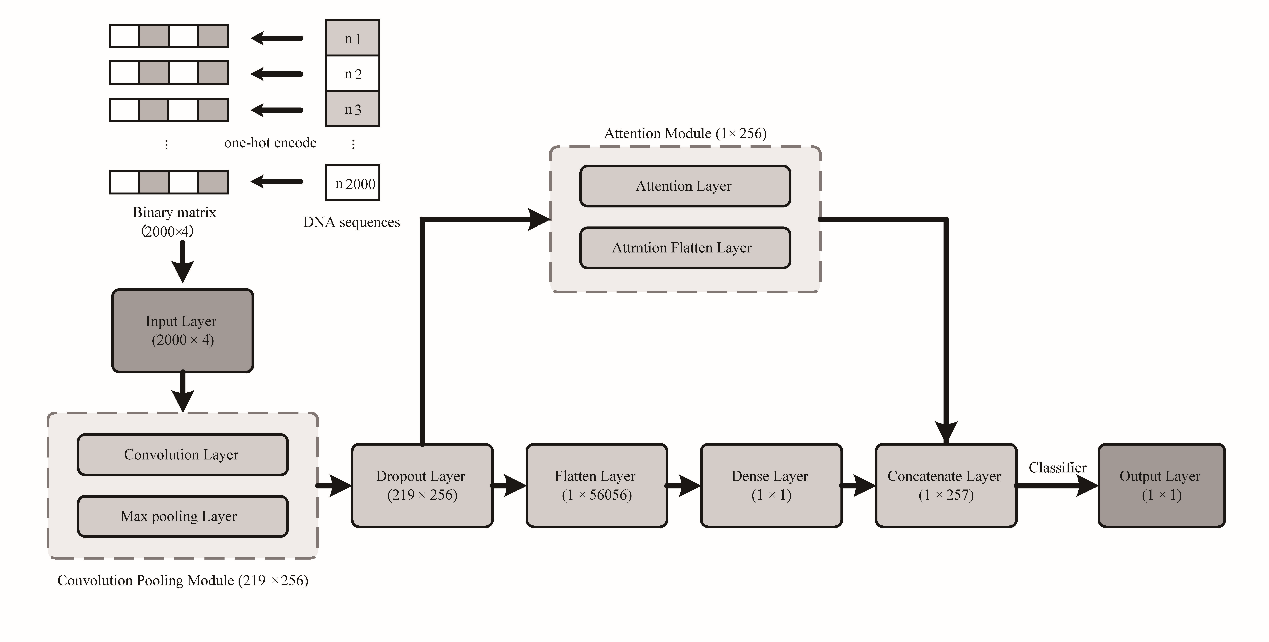 图1 DeepHPV模型结构框架
图1 DeepHPV模型结构框架
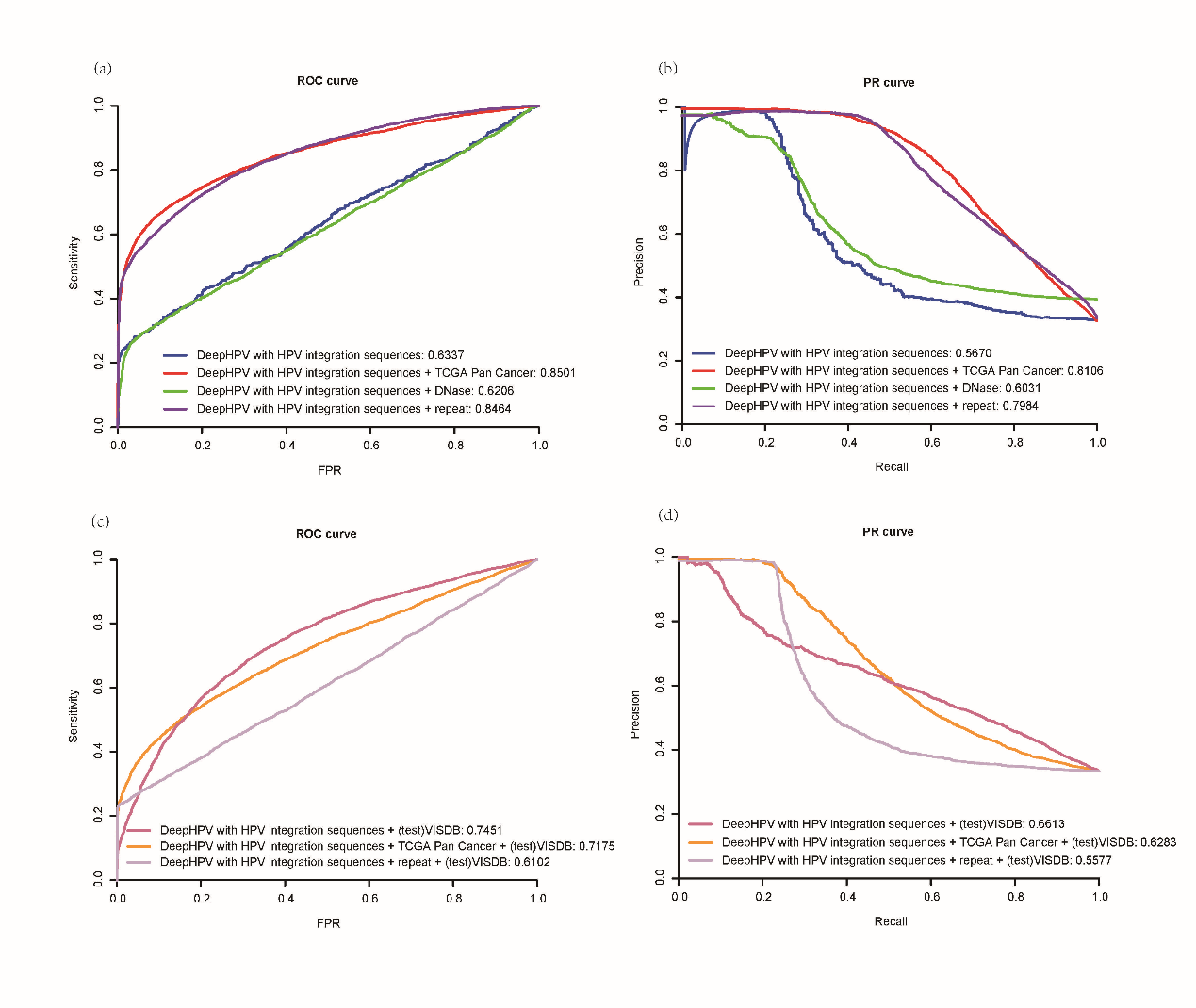
图2 DeepHPV模型预测整合性能结果
本研究进一步地在DeepHPV模型中引入了注意力机制(Attention mechanism),并提取BHLHA15,CHR,COUP-TFII,DMRTA2,E2A,HIC1,INR,NPAS,Nr5a2,RARa,SCL,Snail1,Sox10,Sox3,Sox4等转录因子结合位点(TFBS)附近的注意力富集位点,该结果表明上述相关位点较为脆弱易被HPV整合入侵。
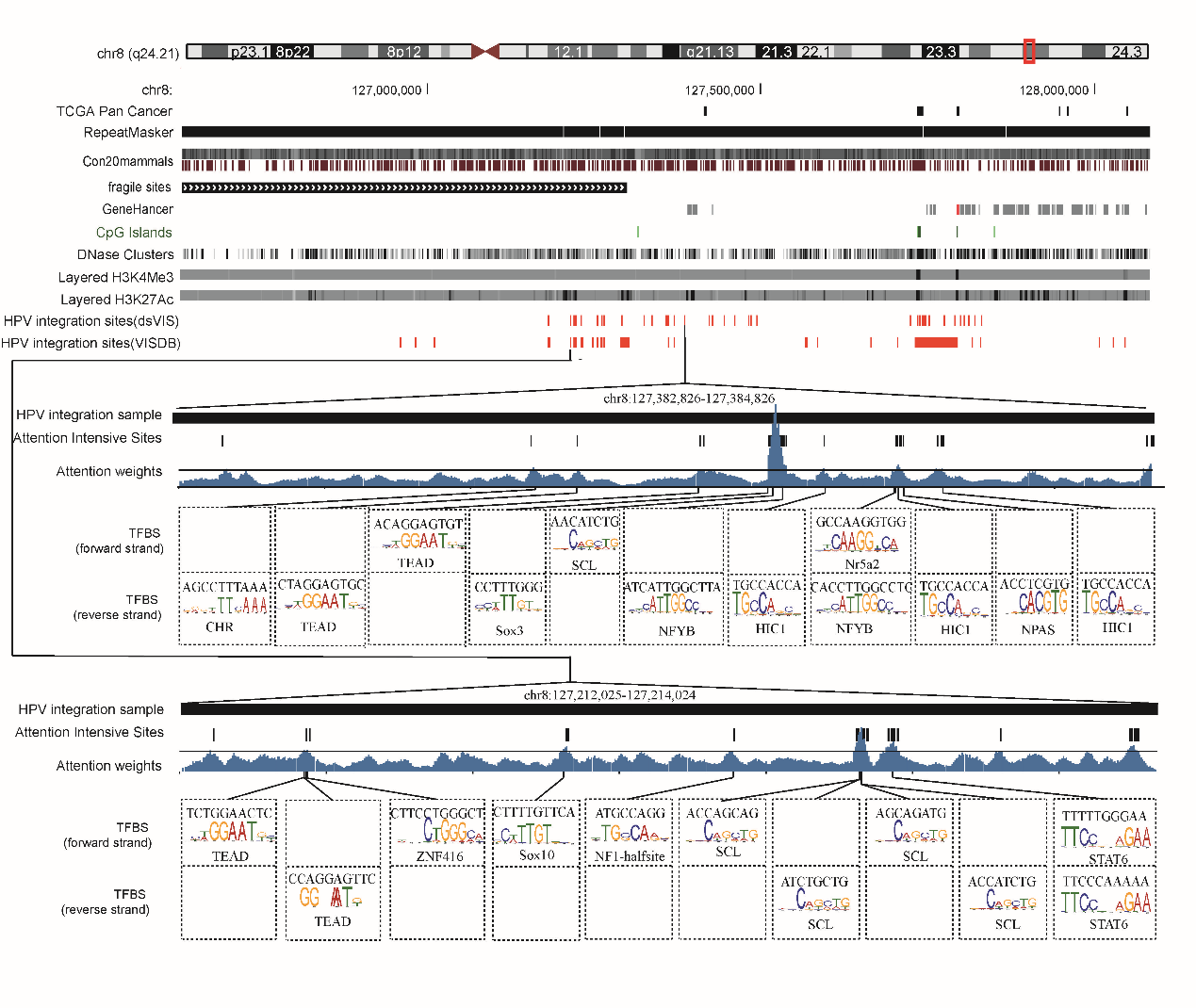
图3 DeepHPV模型注意力机制预测整合富集位点结果
综合以上结果表明本研究中开发的DeepHPV是一个强大且可解释的深度学习模型,尤其对HPV在人类宿主DNA的整合偏好和机制提出了新的方式和见解。
中山大学附属第一医院博士后田瑞为论文第一作者,梁九兴博士与胡争教授为论文共同通讯作者。本项研究受到科技部重点研发计划、国家自然科学基金,广东省重点领域研发计划项目等经费支持。
梁九兴博士是脑科院2018年引进的博士后青年英才,合作导师为国家杰青翁旭初教授。主要研究方向为人工智能在医学工程(癫痫、肿瘤、基因)领域的应用研究。近年作为项目负责人主持国家自然科学基金青年项目,广东省普通高校青年创新人才类项目;作为项目核心骨干参与国家重点研发计划、国家社科重大项目、广东省重点领域研发计划等项目。
原文链接:http://ibrr.scnu.edu.cn/a/20201221/336.html